

Una plataforma de análisis de ARN de código abierto se ha utilizado con éxito en células vegetales por primera vez, un gran avance que podría anunciar una nueva era de investigación fundamental y reforzar los esfuerzos para diseñar plantas de cultivo de biocombustibles y alimentos más eficientes.
La tecnología, llamada Drop-seq, es un método para medir el ARN presente en células individuales, lo que permite a los científicos ver qué genes se están expresando y cómo se relaciona con las funciones específicas de los diferentes tipos de células. Desarrollado en la Escuela de Medicina de Harvard en 2015, el protocolo compartido libremente anteriormente solo se había utilizado en células animales.
“Esto es realmente importante para entender la biología de las plantas”, dijo la investigadora líder Diane Dickel, científica del Laboratorio Nacional Lawrence Berkeley (Berkeley Lab) del Departamento de Energía. “Al igual que los humanos y los ratones, las plantas tienen múltiples tipos de células y tejidos. Pero aprender sobre las plantas a nivel celular es un poco más difícil porque, a diferencia de los animales, las plantas tienen paredes celulares, lo que dificulta la apertura de las células para la genética. estudiar.”
Para muchos de los genes en las plantas, tenemos poca o ninguna comprensión de lo que realmente hacen, explicó Dickel. “Pero al saber exactamente en qué tipo de célula o etapa de desarrollo se expresa un gen específico, podemos comenzar a entender su función. En nuestro estudio, demostramos que Drop-seq puede ayudarnos a hacer esto”.
“También demostramos que puede usar estas tecnologías para comprender cómo responden las plantas a diferentes condiciones ambientales a nivel celular, algo que a muchos biólogos de plantas en Berkeley Lab les interesa porque poder cultivar en condiciones ambientales deficientes, como la sequía, es esencial para nuestra producción continua de alimentos y recursos de biocombustibles “, dijo.
Dickel, quien estudia genómica de mamíferos en la División de Genología Ambiental y Biología de Sistemas de Berkeley Lab, ha estado usando Drop-seq en células animales durante varios años. Una fan inmediata de la facilidad de uso y la eficacia de la plataforma, pronto comenzó a hablar con sus colegas que trabajan en plantas sobre cómo tratar de usarla en células vegetales.
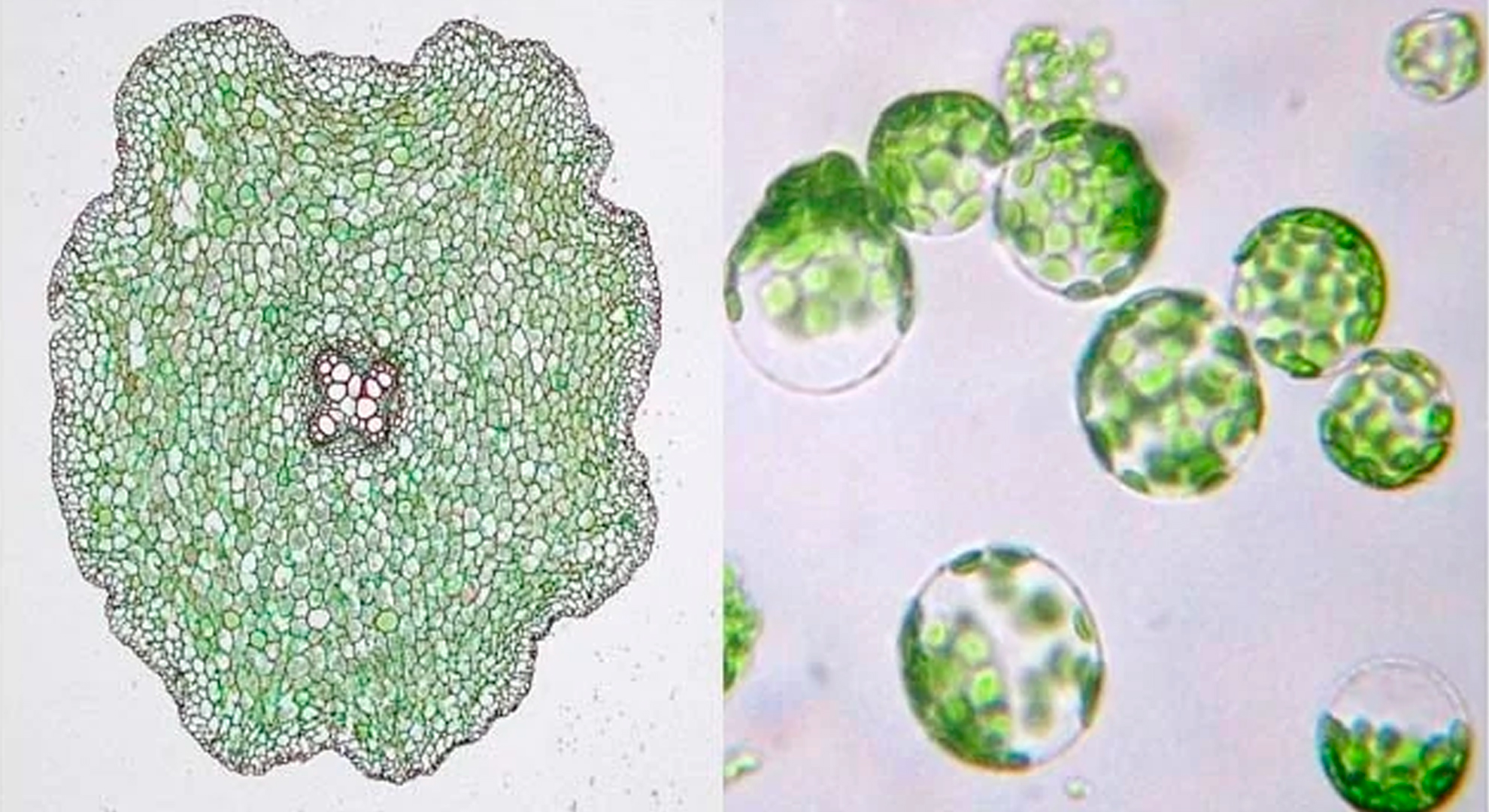
Sin embargo, algunos se mostraron escépticos de que tal proyecto funcionaría con la misma facilidad. En primer lugar, para ejecutar las células vegetales a través de un análisis de secuencias de ARN de una sola célula, deben ser sometidas a protoplastas, lo que significa que deben ser despojadas de sus paredes celulares con un cóctel de enzimas. Este proceso no es fácil porque las células de diferentes especies e incluso diferentes partes de la misma planta requieren cócteles de enzimas únicos.
En segundo lugar, algunos biólogos de plantas han expresado su preocupación de que las células se alteran demasiado significativamente mediante la protoplastia para proporcionar una visión del funcionamiento normal. Y, finalmente, algunas células vegetales son simplemente demasiado grandes para pasarlas por las plataformas existentes de RNA-seq de una sola célula. Estas tecnologías, que surgieron en los últimos cinco años, permiten a los científicos evaluar el ARN dentro de miles de células por ejecución; Los enfoques anteriores solo podían analizar de docenas a cientos de celdas a la vez.
Sin dejarse intimidar por estos desafíos, Dickel y sus colegas en el DOE Joint Genome Institute (JGI) se unieron a investigadores de UC Davis que habían perfeccionado una técnica de protoplastia para el tejido de la raíz de Arabidopsis thaliana (berro de oreja de ratón), una especie de hierba pequeña de floración Que sirve de organismo organismo modelo.
Después de preparar muestras de más de 12,000 células de la raíz de Arabidopsis, el grupo se emocionó cuando el proceso de Drop-seq fue más suave de lo esperado. Sus resultados completos fueron publicados esta semana en Cell Reports .
“Cuando lanzábamos la idea de hacer esto en las plantas, la gente mostraba una lista de razones por las cuales no funcionaría”, dijo Dickel. “Y diríamos, ‘está bien, pero intentémoslo y veamos si funciona’. Y luego realmente funcionó. Nos sorprendió sinceramente lo sencillo que realmente terminó siendo”.
La naturaleza de código abierto de la tecnología Drop-seq fue crítica para el éxito de este proyecto, según el coautor Benjamin Cole, un científico de genómica de plantas en JGI. Debido a que Drop-seq es económico y utiliza componentes fáciles de ensamblar, les brindó a los investigadores un medio de bajo riesgo y bajo costo para experimentar. Ya se está construyendo una ola de interés. En el tiempo previo a la publicación de su artículo, Dickel y sus colegas comenzaron a recibir solicitudes, de parte de otros científicos en Berkeley Lab, JGI, y más allá, para obtener consejos sobre cómo adaptar la plataforma para otros proyectos.
“Cuando hablé por primera vez con Diane para probar el Drop-seq en plantas, reconocí el enorme potencial, pero pensé que sería difícil separar las células de la planta lo suficientemente rápido como para obtener datos útiles”, dijo John Vogel, científico líder de genómica funcional de plantas en JGI. “Me sorprendió ver lo bien que funcionó y lo mucho que pudieron aprender de su experimento inicial. Esta técnica va a ser un cambio de juego para los biólogos de plantas porque nos permite explorar la expresión genética sin moler órganos de plantas enteras, y los resultados no se confunden con las señales de los pocos tipos de células más comunes “.
Los autores anticipan que la plataforma y otras tecnologías similares de RNA-seq, eventualmente se convertirán en una rutina en las investigaciones de plantas. El principal obstáculo, señaló Dickel, será el desarrollo de métodos de protoplasto para cada planta de interés del proyecto.
“Parte de la misión de Berkeley Lab es comprender mejor cómo responden las plantas a las condiciones ambientales cambiantes, y cómo podemos aplicar este entendimiento para utilizar mejor las plantas para la bioenergía”, señaló la primera autora Christine Shulse, actualmente afiliada a JGI. “En este trabajo, generamos un mapa de expresión génica en tipos de células individuales de una especie de planta en dos condiciones ambientales, lo que es un primer paso importante”.
Este artículo ha sido reeditado a partir de materiales proporcionados por el Laboratorio de Berkeley.. Nota: el material puede haber sido editado por longitud y contenido. Para más información, contactar con la fuente citada.
Referencia: Shulse et al. 2019. Perfilado de alto rendimiento de transcriptomas de células individuales de tipos de células vegetales. Informes celulares. DOI:
https: //doi.org/10.1016/j.celrep.2019.04.054.
Fuente: www.technologynetworks.com