Los hospitales de la UC y la U de Chile ya están prescribiendo el medicamento a personas que cumplen con determinados requisitos, mientras que la llegada al sistema público comenzaría durante la primera quincena de junio en dos hospitales. Y ojo: no se debe reemplazar el uso del condón, ya que no es 100% eficaz.
“Les cuento que acabo de empezar a tomar PrEP: profilaxis preexposición. ¿Qué significa? Es un tratamiento preventivo para disminuir la probabilidad de adquirir VIH. No es 100% eficaz, por lo que no debe reemplazar el uso del condón”.
Este fue el mensaje que publicó esta mañana en sus historias de Instagram el ex presidente de la Fundación Iguales, Luis Larraín, al comunicar que se había incorporado a uno de los programas que ya están prescribiendo este medicamento, que en otros países ha sido clave para la disminución de casos positivos de VIH.
“Ya tengo una enfermedad crónica. Agregar otra enfermedad crónica como el VIH me complicaría bastante la vida, por lo que quise tomar todos los resguardos para disminuir al máximo la probabilidad de tenerlo”, explica a T13 Larraín, quien padece de una enfermedad renal crónica.
Fue durante 2018 que el Ministerio de Salud anunció la incorporación del PrEP al Plan Nacional contra el VIH/Sida. Una medida que buscaba enfrentar el explosivo aumento de casos positivos: solo entre enero y diciembre de se registraron 6.948 nuevos casos de VIH en Chile, 1.132 más que en 2017. Esto representa un alza de más del 96% desde el años 2010.
¿Cómo acceder a la pastilla? ¿Cómo funciona? ¿Cuándo será incorporada al sistema público?
Una pastilla al día, y solo para personas que cumplen con requisitos
La profilaxis preexposición (PrEP), consiste en el uso de antiretrovirales para reducir el riesgo de adquirir una infección por VIH. Su eficacia se ubica en torno al 90% y ha sido demostrada en diversos estudios clínicos.
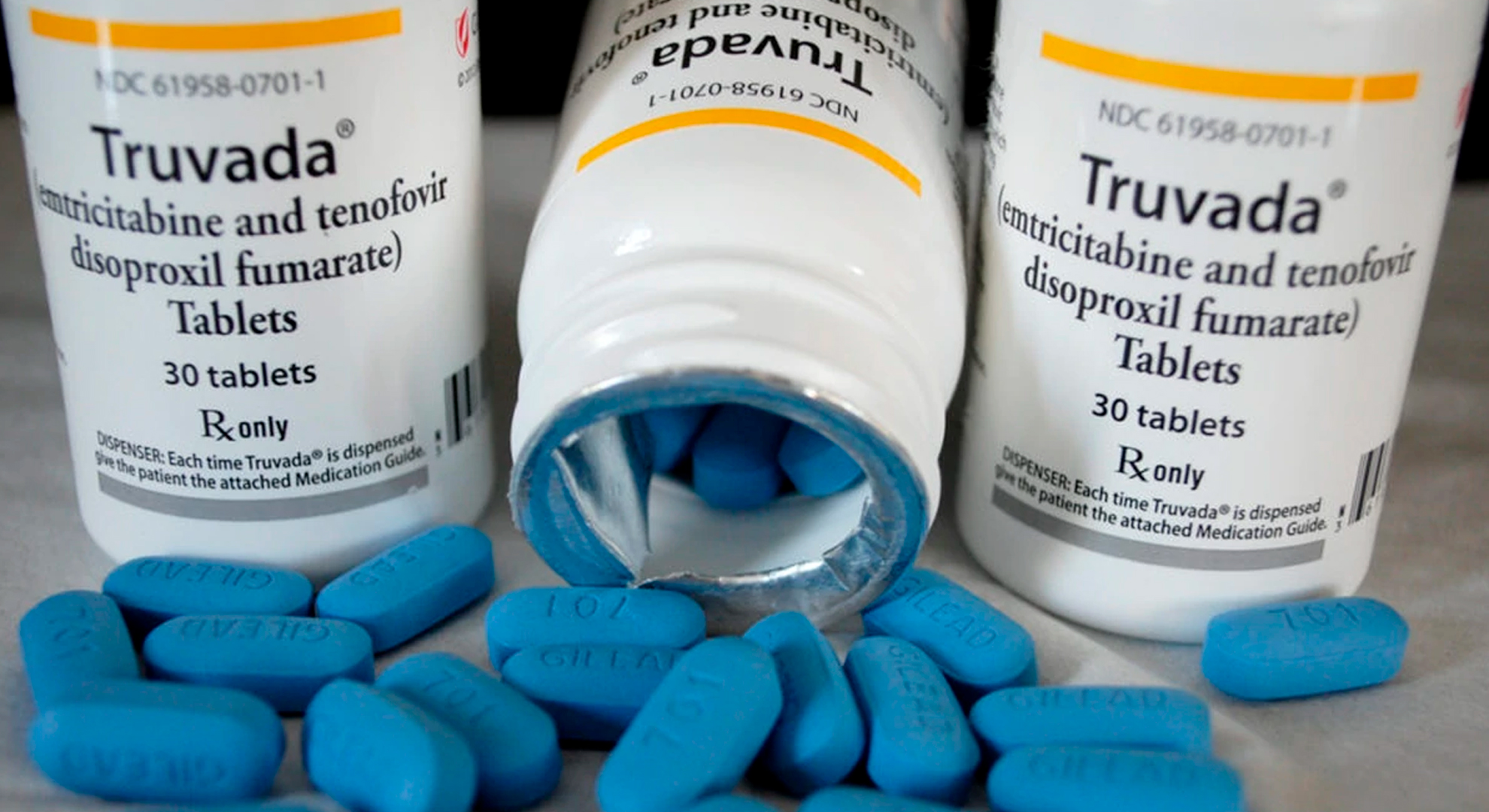
Se trata Truvada, una pastilla que combina los compuestos tenofovir y emtricitabina, dos antiretrovirales que impiden la multiplicación del virus y que éste se incorpore en las células. Y se toma una vez al día, siempre a la misma hora, según las preferencias del usuario.
El PrEP ha sido recomendado por la OMS y Onusida y ya se utiliza en EE.UU., Australia y Europa. Brasil, México y Perú son algunos países de sudamérica que ya lo han incorporado en sus estrategias de prevención.
Los Hospitales Clínicos de la Universidad Católica y de la Universidad de Chile llevan algunos meses prescribiendo este medicamento a personas que cumplen ciertos requisitos, a través de un convenio que les permite adquirirlo a un precio rebajado en farmacias. Mientras el precio normal del Truvada supera los 400 mil pesos, con este programa pueden obtenerlo a 69 mil. Eso sí, hay versiones bioequivalentes a menor precio.
“El programa sigue la misma línea de lo que se hace en otros centros del mundo y lo que recomienda la OMS. Y se recomienda a personas que no tienen VIH y que están en riesgo de adquirirlo”, explica el doctor Carlos Pérez, infectólogo de la Facultad de Medicina y la Red de Salud UC Christus.
¿Quiénes pueden acceder al PrEP?
- Personas que tienen relaciones sexuales no protegidas
- Personas que tienen más de una pareja sexual
- Personas que han tenido enfermedades o infecciones de transmisión sexual
- Personas que tienen una pareja portadora VIH que no está recibiendo tratamiento o que está recién iniciándolo.
El doctor Alejandro Afani, inmunólogo y director del Centro de VIH del Hospital Clínico de la Universidad de Chile, detalla que “en los dos últimos meses ha tenido bastantes requerimientos, hay muchos jóvenes interesados. Y a los que han acudido a mí siempre les hago una encuesta, porque esto no es para todo el mundo”.
Tanto en la UC como en la U. de Chile se les realiza una encuesta a los interesados, para ver si forman parte de los grupos de riesgo. En caso de ser así, se les ordena una serie de exámenes, entre ellos el test de VIH. El primer paso es solicitar una hora con alguno de los infectólogos de los recintos.
En caso de cumplir con los requisitos, se les receta el PrEP, con una instrucción expresa: controles cada tres meses, para descartar la presencia del virus. “Es importante que las personas sepan que es muy importante tomarlo diariamente porque la adherencia está directamente vinculada con los resultados”, agrega.
Junto con esto, se debe evaluar la eventual presencia de efectos secundarios, como nauseas, erupciones cutáneas, dolor abdominal, dolor de cabeza, depresión, fatiga y trastornos del sueño, entre otros.
Los doctores Pérez y Afani son enfáticos en que la eficacia no es de un 100%, por lo que sigue siendo necesario el uso de condón. Además, el PrEP no previene otras infecciones de transmisión sexual (ITS), como la gonorrea, sífilis, clamidia, entre otras.
De hecho, en otros países donde se ha implementado, se ha visto que aunque se han detenido o han bajado considerablemente los nuevos casos de VIH, las tasas de contagio de otras ITS se han mantenido.
¿Cuándo se incorporará en la salud pública?
Según detallan desde el Minsal, la estrategia de prevención a través del PrEP debutará durante la primera quincena de junio en dos establecimientos de atención secundaria (hospitales) que se informarán oportunamente. La llegada a la atención primaria y otros hospitales se hará de forma gradual.

Al igual que la UC y la U. de Chile, el programa tiene como requisito que las personas no sean portadoras de VIH y que cumplan con requisitos como haber tenido relaciones sin protección, más de una pareja en los últimos seis meses, haber tenido alguna ITS, o ejercer el comercio sexual.
Se estima que cerca de 5 mil personas podrían acceder al tratamiento de manera gratuita a través del sistema público.
Junto con esto, será necesario que los recintos asistenciales cuenten con acceso a tamizaje para el VIH y técnicas de detección de sífilis, clamidia y gonorrea. Además de los recursos humanos para evaluar el comportamiento de los pacientes y realizar la debida orientación para la adherencia del medicamento y sobre cómo tener relaciones sexuales seguras y protegidas, enfatizando que el PrEP previene el VIH y no otras ITS.
Adicionalmente, se requerirá de hacer un chequeo cada tres meses a las personas que reciban la pastilla.
“Lo importante es masificarlo rápidamente, en especial en las regiones donde tenemos una alta prevalencia, particularmente en la zona norte. Me parece bien que se active este primer semestre, pero en el corto plazo hay que masificarlo”, señala el doctor Afani.
El especialista destaca la importancia de la incorporación del PrEP a la salud pública de manera gratuita, porque actualmente “solo algunas personas tienen acceso”.